Язык программирования R активно применяется для анализа данных в бизнесе и науке. Это важный и очень популярный инструмент науки о данных. Его предпочитают многие статистики и дата-сайентисты, которые часто работают с большими объемами данных и сложными статистическими задачами.
Главная роль здесь отводится памяти и времени выполнения. Для максимальной производительности нужен эффективный код. Напишите его в своем следующем проекте на R, следуя этим семи рекомендациям.
Рекомендация 1. Выполняйте профилирование кода
Дата-сайентисты часто стремятся оптимизировать код, сделать его быстрее. Но если довериться интуиции, можно оптимизировать не те части кода, потеряв время и силы. Решение — профилирование. Код оптимизируется, только когда известны медленные его части, которые находят при профилировании.
Rprof() — встроенный инструмент профилирования кода. Использовать его напрямую не очень удобно, поэтому рекомендуем пакет profvis для визуализирования данных профилирования кода из Rprof(). Пакет устанавливается консольной командой:
install.packages("profvis")
Вот пример профилирования кода:
library("profvis")
profvis({
y <- 0
for (i in 1:10000) {
y <- c(y,i)
}
})
Запустив этот код в RStudio, получаем:
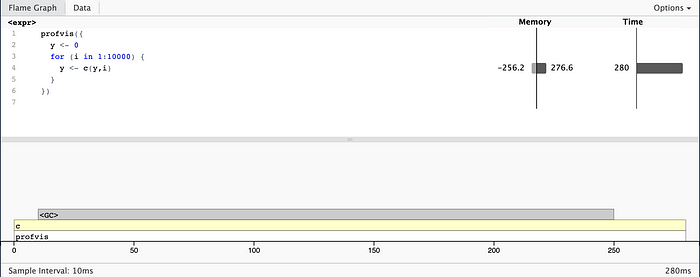
Вверху показан код на R с гистограммами памяти и времени выполнения каждой его строки. Здесь обрисовываются возможные проблемы кода, но без точной причины. В столбце memory баром слева показано, сколько памяти в Мб для каждого вызова выделено, справа — сколько освобождено. В столбце time дано время выполнения каждой строки в мс, например строка 4 — 280 мс.
Внизу показан Flame Graph с полным стеком вызовов. Здесь дается вся их последовательность. Наводя курсор на отдельные вызовы, получаем данные о них. Посмотрите: сборщиком мусора <GC> тратится много времени. Но почему? В столбце memory наблюдается повышенная потребность в памяти: много ее выделяется и освобождается в строке 4. Каждой итерацией создается очередная копия y, отсюда повышенное потребление памяти. Избегайте таких задач копирования/изменения.
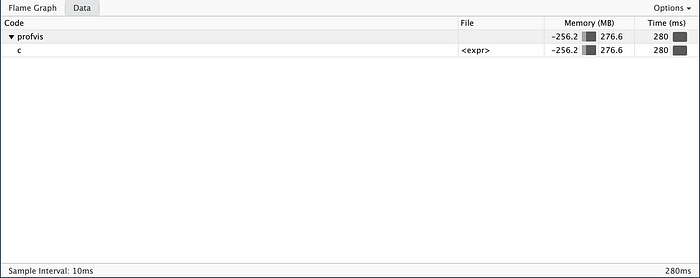
Еще имеется вкладка Data с перечнем всех вызовов, особенно удобная для сложных вложенных вызовов:
Подробнее о provis — на странице Github.
Рекомендация 2. Векторизируйте код
Векторизация — это не только уход от циклов for(), но и замена скаляров векторами. Векторизация очень важна для ускорения кода на R. В векторизованных функциях применяют циклы, написанные не на R, а на C: у них меньше накладных расходов и они намного быстрее. Векторизация — это поиск имеющейся функции на R, которая реализована на C и точно соответствует задаче. Векторизованные матричные функции rowSums(), colSums(), rowMeans() и colMeans() удобны для ускорения кода на R и всегда выполняются быстрее функции apply().
Для измерения времени выполнения используется пакет microbenchmark на R. Чтобы минимизировать накладные расходы, все выражения в нем вычисляются на C, на выходе выдается сводка статистических показателей. Пакет microbenchmark устанавливается консольной командой:
install.packages("microbenchmark")
Сравним время выполнения apply() и colMeans():
install.packages("microbenchmark")
library("microbenchmark")
data.frame <- data.frame (a = 1:10000, b = rnorm(10000))
microbenchmark(times=100, unit="ms", apply(data.frame, 2, mean), colMeans(data.frame))
# пример консольного вывода:
# Единица измерения: миллисекунды
# expr min lq mean median uq max neval
# apply(data.frame, 2, mean) 0.439540 0.5171600 0.5695391 0.5310695 0.6166295 0.884585 100
# colMeans(data.frame) 0.183741 0.1898915 0.2045514 0.1948790 0.2117390 0.287782 100
В обоих случаях вычисляем среднее значение по каждому столбцу фрейма данных. Для надежности результата делаем 100 запусков (times=10), используя пакет microbenchmark. Итог: функция colMeans() примерно в три раза быстрее.
Хотите знать больше о векторизации? Рекомендуем онлайн-книгу R Advanced (Eng).
Рекомендация 3. Матрицы вместо фреймов данных
Матрицы похожи на фреймы данных: это двумерные объекты, работа некоторых функций одинакова. Разница: все элементы матрицы должны иметь один тип.
Матрицы часто используются для статистических вычислений. Например, в функции lm() входные данные преобразуются в матрицу. Затем вычисляются результаты. В целом матрицы быстрее фреймов данных.
Рассмотрим различия между ними во времени выполнения:
library("microbenchmark")
matrix = matrix (c(1, 2, 3, 4), nrow = 2, ncol = 2, byrow = 1)
data.frame <- data.frame (a = c(1, 3), b = c(2, 4))
microbenchmark(times=100, unit="ms", matrix[1,], data.frame[1,])
# пример консольного вывода:
# Единица измерения: миллисекунды
# expr min lq mean median uq max neval
# matrix[1, ] 0.000499 0.0005750 0.00123873 0.0009255 0.001029 0.019359 100
# data.frame[1, ] 0.028408 0.0299015 0.03756505 0.0308530 0.032050 0.220701 100
Чтобы получить статистически значимую оценку, делаем 100 запусков с пакетом microbenchmark. Очевидно, что доступ к первой строке у матрицы примерно в 30 раз быстрее, чем у фрейма данных. Матрица значительно быстрее, поэтому предпочтительнее фрейма данных.
Рекомендация 4. is.na() и anyNA
Имеется две функции для проверки пропущенных значений в векторе: is.na() и anyNA(). Тестируем, какая функция выполняется быстрее:
library("microbenchmark")
x <- c(1, 2, NA, 4, 5, 6, 7)
microbenchmark(times=100, unit="ms", anyNA(x), any(is.na(x)))
# пример консольного вывода:
# Единица измерения: миллисекунды
# expr min lq mean median uq max neval
# anyNA(x) 0.000145 0.000149 0.00017247 0.000155 0.000182 0.000895 100
# any(is.na(x)) 0.000349 0.000362 0.00063562 0.000386 0.000393 0.022684 100
anyNA() в среднем значительно быстрее, чем is.na(), по возможности используйте ее.
Рекомендация 5. if() … else() вместо ifelse()
if() ... else() — стандартная функция потока управления, а ifelse() удобнее для пользователя.
Схема работы Ifelse():
# тест: условие, если_да: условие true, если_нет: условие false
ifelse(test, if_yes, if_no)
С точки зрения многих программистов, ifelse() понятнее многострочной альтернативы. Недостаток: ifelse() не столь эффективна в вычислительном отношении. В этом тесте производительности if() ... else() выполняется более чем в 20 раз быстрее:
library("microbenchmark")
if.func <- function(x){
for (i in 1:1000) {
if (x < 0) {
"negative"
} else {
"positive"
}
}
}
ifelse.func <- function(x){
for (i in 1:1000) {
ifelse(x < 0, "negative", "positive")
}
}
microbenchmark(times=100, unit="ms", if.func(7), ifelse.func(7))
# пример консольного вывода:
# Единица измерения: миллисекунды
# expr min lq mean median uq max neval
# if.func(7) 0.020694 0.020992 0.05181552 0.021463 0.0218635 3.000396 100
# ifelse.func(7) 1.040493 1.080493 1.27615668 1.163353 1.2308815 7.754153 100
Избегайте ifelse() в сложных циклах, иначе работа программы значительно замедлится.
Рекомендация 6. Параллельные вычисления
Большинство процессоров многоядерные, то есть с параллельной обработкой задач или параллельными вычислениями, которые возможны в приложениях на R благодаря предустановленному в базовом R пакету parallel. Этой командой загружаем его и посмотрим, сколько ядер у компьютера:
library("parallel")
no_of_cores = detectCores()
print(no_of_cores)
# пример консольного вывода:
# [1] 8
Параллельная обработка данных идеальна для моделирования методом Монте-Карло. Реализация модели воспроизводится каждым ядром независимо, в конце подводятся итоги.
Следующий пример взят из онлайн-книги Efficient R Programming (Эффективное программирование на R). Сначала устанавливаем пакет devtools. С его помощью загружаем с GitHub пакет efficient. Для этого вводим в консоли RStudio следующие команды:
install.packages("devtools")
library("devtools")
devtools::install_github("csgillespie/efficient", args = "--with-keep.source")
В пакете efficient имеется функция snakes_ladders(), которой моделируется одиночная игра «Змейки и лестницы». Применим это моделирование для измерения времени выполнения функции sapply() и ее распараллеленного варианта parSapply():
library("parallel")
library("microbenchmark")
library("efficient")
N = 10^4
cl = makeCluster(4)
microbenchmark(times=100, unit="ms", sapply(1:N, snakes_ladders), parSapply(cl, 1:N, snakes_ladders))
stopCluster(cl)
# пример консольного вывода:
# Единица измерения: миллисекунды
# expr min lq mean median uq max neval
# sapply(1:N, snakes_ladders) 3610.745 3794.694 4093.691 3957.686 4253.681 6405.910 100
# parSapply(cl, 1:N, snakes_ladders) 923.875 1028.075 1149.346 1096.950 1240.657 2140.989 100
Моделирование с parSapply() в среднем примерно в 3,5 раза быстрее, чем с sapply(). Эта рекомендация быстро интегрируется в имеющийся проект на R.
Рекомендация 7. Интерфейс сопряжения R с другими языками
Бывают ситуации, где код на R просто медленный и никакими приемами не убыстряется. Перепишите его на другой язык — для них в R имеются интерфейсы в виде R-пакетов, например Rcpp и rJava. Легко написать код на C++, особенно если имеется опыт разработки ПО, и использовать этот код на R.
Сначала устанавливаем Rcpp:
install.packages("Rcpp")
Продемонстрируем применяемый подход:
library("Rcpp")
cppFunction('
double sub_cpp(double x, double y) {
double value = x - y;
return value;
}
')
result <- sub_cpp(142.7, 42.7)
print(result)
# консольный вывод:
# [1] 100
C++ — мощный, самый подходящий для ускорения кода язык. Для очень сложных вычислений рекомендуем использовать код на нем.
Заключение
Из статьи мы узнали, как анализируется и ускоряется код на R.
- Анализ кода поддерживается пакетом provis.
- Применяются векторизованные функции
rowSums(),colSums(),rowMeans()иcolMeans(). - Где возможно, вместо фреймов данных используются матрицы.
- Пропущенные значения в векторе проверяются с помощью
anyNA(), а неis.na(). - Код на R ускоряется использованием
if() ... else()вместоifelse(). - Для сложного моделирования применяются распараллеленные функции пакета parallel.
- Максимальная производительность сложных частей кода достигается использованием пакета Rcpp.
Читайте также:
- Создание анимированной пузырьковой диаграммы Ханса Рослинга на языке R
- Создание API в R при помощи Plumber
- Три библиотеки R, которые должен знать каждый специалист по данным
Читайте нас в Telegram, VK и Дзен
Перевод статьи Janik Tinz: Boost your R Skills to the next level